Биоинформатики ИТМО разработали сервис для анализа клеточного метаболизма — биохимических реакций, отвечающих за жизнедеятельность клеток, — Shiny GATOM. В отличие от аналогов предложенный инструмент рассматривает клеточные процессы на самом глубоком уровне — не только на уровне веществ и генов, но и на уровне атомов. Это облегчает интерпретацию результатов. Сервис будет полезен для решения задач в биологии и медицине — например, он может помочь в разработке лекарств против аутоиммунных заболеваний и рака.
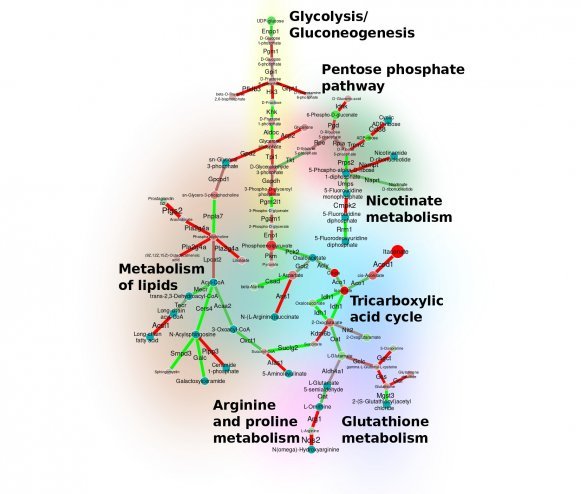
Граф процесса расщепления глюкозы (гликолиза и глюконеогенеза)
© ИТМО
Метаболизм в клетках играет важную роль в регуляции многих биологических процессов, в том числе работы иммунной системы. Глубинное изучение метаболизма помогает лучше понять, как в живых организмах протекают аутоиммунные заболевания или развивается рак, а также создавать новые методы лечения этих болезней. Однако существующие решения позволяют разобрать лишь ограниченный набор стандартных химических реакций. Для нетривиальных задач — например, для анализа сбоев и «поломок» в клеточном метаболизме — нужны более сложные программные продукты. Кроме того, необходимы инструменты, которые смогут представлять детальную информацию не только о метаболическом пути в целом, но и об его отдельно взятых участках.
Работа над новым решением началась еще в 2016 году — тогда ученым из ИТМО совместно с зарубежными коллегами удалось разработать веб-сервис, который обнаруживает связи между изменениями в метаболизме и генах. Сейчас это уже полноценная программа, которую можно установить на компьютер.
«Принцип работы с программой достаточно простой. Исследователь загружает в нее таблицу с данными по метаболитам (простым низкомолекулярным веществам, вовлеченным в обмен веществ) и активности генов, например, раковой клетки. Программа сравнивает эти данные с базами KEGG и Rhea, в которых описано большинство биологических процессов в стандартном состоянии. Результат выдается в виде некой карты, графа, где наглядно представлен путь превращения веществ и видны связи между ними. Вершины этого графа — вещества; линии между ними (“ребра”) — реакции. Причем алгоритм сам выделяет цветом те метаболиты и кодирующие их гены, на которые нужно обратить внимание. На это все уходит около минуты», — рассказывает Алексей Сергушичев, директор научно-образовательного центра геномного разнообразия и руководитель фронтирной лаборатории «Вычислительные методы для системной биологии» в ИТМО.
За последние несколько лет функционал сервиса расширился — он теперь умеет анализировать биохимические реакции не только на уровне веществ, но и на уровне атомов. Это позволяет лучше структурировать граф и облегчает интерпретацию результатов. Однако в графе с превращениями отдельных атомов усложняется поиск подграфов с наиболее выраженными изменениями, ведь атомы могут встречаться сразу в нескольких веществах, а значит, количество рассматриваемых реакций увеличивается в несколько раз. Ученым удалось решить и эту проблему — сервис сам убирает ненужные повторы и оставляет лишь «ребра» и вершины с положительным весом, то есть высоким показателем значимости.
«Еще одно важное обновление — возможность работать с липидомными данными (информацией о жирах и их производных). Изучая их, можно, например, узнать, как устроены разные отделы головного мозга, где разнообразие жирового состава играет большую роль. Липидомика — относительно новое, но активно развивающееся направление, и программ, которые умеют анализировать метаболические липидные процессы, практически нет. Однако реакции с липидами уже хорошо описаны в базе Rhea — теперь она также является частью алгоритма GATOM», — объяснила Мария Емельянова, первый автор статьи, программист научно-образовательного центра геномного разнообразия ИТМО.
Благодаря разработанному вычислительному методу авторы уже смогли связать развитие болезни Альцгеймера с «поломкой» в гене TREM2 и показали, что снизить темпы роста опухоли можно за счет замедления определенных процессов метаболизма в раковых клетках. В дальнейших планах ученых — добавить новые базы данных с реакциями, а также повысить эффективность и скорость работы с алгоритмами. Исследование реализовано в рамках программы «Приоритет 2030» Минобрнауки России.
Статья опубликована в журнале Nucleic Acids Research
Информация взята с портала «Научная Россия» (https://scientificrussia.ru/)
Источник: sci-dig.ru